DNA dizileme
DNA dizilemesi, bir DNA molekülündeki nükleotit bazlarının (adenin, guanin, sitozin ve timin) sırasının belirlenmesidir.
DNA dizilerinin bilinmesi temel biyoloji, biyoteknoloji, adli bilim, tıbbi tanı koyma gibi pek çok sahada vazgeçilmez hâle gelmiştir. DNA dizilemesi biyolojik araştırma ve keşifleri çok hızlandırmıştır. Modern DNA dizileme teknolojilerin mümkün kıldığı hızlı DNA dizileme sayesinde İnsan Genom Projesi'nde insan genomunun dizilenebilmiştir. Benzer projelerle pek çok hayvan, bitki ve mikrop genomunun tam dizisi üretilebilmiştir.
İlk DNA dizileri 1970'lerin başlarında üniversite araştırmacıları tarafından iki-boyutlu kromatografiye dayanan zahmetli yöntemlerle elde edilmiştir. Otomatik analizle çalışan boya-tabanlı dizileme yöntemlerinin gelişimiyle[1] DNA dizilemesi çok daha kolaylaşmış ve birkaç büyüklük mertebesi hızlanmıştır.[2]
Tarih
RNA dizilemesi nükleotit dizilemesinin en erken biçimlerinden oldu. RNA dizilemesinin en önemli aşamaları, Gent Üniversitesi'nden (Gent, Belçika) Walter Fiers ve arkadaşlarının, 1972'de Bakteriyofaj MS2'ye ait bir genin,[3] daha sonra 1976'da ise aynı bakteriyofajın tüm genomunun[4] dizisi olmuştur.
1970'lerin başlarında hızlı DNA dizileme yöntemlerinin geliştirilene kadar, DNA dizilemesi için çeşitli zahmetli yöntemler kullanılmaktaydı.[5][6] Örneğin, 1973'te Gilbert ve Maxam, dolaşan benek analiz (wandering-spot analysis) olarak adlandırılan bir yöntemle 24 nükleotitin dizisini yayımladılar.[7]
Sanger ve arkadaşlarının 1975'te geliştirdiği zincir sonlandırma yöntemi kısa sürede en tercih edilen yöntem oldu, göreceli olarak daha kolay ve güvenilir olmasından dolayı.[8][9]
Maxam - Gilbert dizilemesi
1976-1977'de Harvard Üniversitesi'nden Allan Maxam and Walter Gilbert, DNA'nın kimyasal modifikasyonu ve ardından onun spesifik bazlarda kesilmesi esasına dayanan bir DNA dizileme yöntemi geliştirdi.[5] Maxam ve Gilbert kimyasal dizileme yöntemi hakkındaki makale, Sanger ve Coulson'un artı-eksi dizilemesi hakkındaki makalesinden iki yıl daha sonra yayınlamasına rağmen,[8][10] Maxam-Gilbert dizilemesi daha popüler oldu. Bunun nedeni, Maxam-Gilbert yönteminde saflaştırılmış DNA'nın doğrudan dizilenebilmesi, buna karşın ilk Sanger yönteminde tek iplikli DNA üretilebilmesi için okunacak her DNA'nın ayrıca klonlanmasının gerekmesiydi. Ancak, zincir sonladırma yönteminin zaman içinde iyileştirilmesiyle Maxam-Gilbert dizilemesi gözden düştü, zira teknik karmaşıklığı onun standart moleküler biyoloji kitlerinde kullanılmasına olanak vermiyordu, ayrıca zararlı kimyasallara gerek gösteriyordu ve ölçeklenmesinde zordu.[11]
Bu yöntem DNA'nın 5' ucunun radyoaktif olarak işaretlenmesini (bu tipik olarak gamma-32P ATP kullanılan bir kinaz reaksiyonuydu) ve sonra dizilenecek DNA parçasının saflaştırılmasını gerektirir. Dört farklı kimyasal reaksiyonda (G, A+G, C, C+T) uygulanan kimyasal işlemlerle, moleküllerin ufak bir kısmında, DNA'yı oluşturan dört nükleotit bazdan biri veya ikisinde kesikler meydana gelir. Örneğin, pürinler formik asitle depürine edilir, guaninler (ve bir miktar adeninler de) dimetil sülfat ile metillenir, pirimidinler (C+T) hidrazin ile metillenir. Sadece-C reaksiyonunda, hidrazin reaksiyonuna tuz (sodyum klorür) eklenmesi timinin metillenmesini inhibe eder. Modifiye olmuş DNA'lar sonra sıcak piperidin ile modifiye olmuş bazların poziyonunda kesilir. Modifikasyon yapan kimyasalların konsantrasyonu ayarlanarak DNA molekülü başına ortalama bir modifikasyon olması sağlanır. Böylece bir seri işaretlenmiş DNA parçası elde edilir, bu parçaların bir ucunda radyoaktif işaret, öbür ucunda ise ilk "kesik" yeri olur. Dört reaksiyonda meydana gelen parçalar, denatüran akrilamid jel içinde yan yana elektroforezle ayrıştırılır. Parçaları görselleştirmek için jel bir röntgen filminin üzerine konur, böylece her bir radyoaktif DNA parşasının bulunduğu yere karşılık gelen noktada fotoğraf filmi kararır. Filmde meydan gelen bant serilerinden DNA dizisi çıkarılabilir.[12]
"Kimyasal dizileme" olarak bilinen bu yöntem daha sonradan DNA'ya bağlanan proteinlerin DNA'ya bağlanma yerlerini haritalamak için kullanılan Metilasyon Enterferans Ölçümü'nün temelini oluşturmuştur.[13]
Zincir sonlandırma yöntemleri
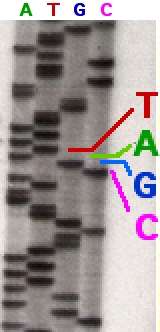
Zincir sonlandırma yöntemi (veya Sanger yöntemi, onu geliştiren Frederick Sanger'e atfen) Maxam ve Gilbert yöntemine kıyasla daha verimli olduğu, daha az toksik kimyasal ve radyoaktivite gerektirdiği için kısa sürede hızla yaygınlaştı. Sanger yönteminin ana ilkesi zincir sonladırıcı olarak dideoksinükleotit trifosfatlar (ddNTP'ler) kullanılmasıdır.
Klasik zincir sonladırma yöntemi için tek iplikli bir DNA kalıp, bir DNA primeri, bir DNA polimeraz, normal deoksinükleotitfosfatlar (dNTP'ler) ve DNA zincir büyümesini sonladıran modifiye edilmiş nükleotitler (dideoksiNTP'ler, veya kısaca ddNTP'ler) kullanılır. Bu ddNTP'ler otomatik dizileme makinalarında otomatik olarak tespit edilebilmek için radyokatif veya floresan olarak işaretlenir. DNA numunesi dört ayrı dizileme reaksiyonu için paylaştırılır, bunlarda ortak olarak standart deoksinükleotitler (dATP, dGTP, dCTP ve dTTP) ve DNA polimeraz bulunur. Her reaksiyona dört dideoksinükleotit'ten bir tanesi (ddATP, ddGTP, ddCTP, veya ddTTP) eklenir. İki nükleotit arasında bir fosfodiester bağı oluşması için gerekli olan 3'-OH, dideoksinükleotitlerde bulunmadığı için bunları içeren bir DNA zinciri daha fazla uzayamayaz. Bu nedenle, çeşitli uzunluklarda DNA zincirlerinin oluşumuyla reaksiyon sona erer.
Yeni sentezlenen ve işaretlenen DNA parçaları ısıtılarak denatüre edilir ve jel elektroforeziyle büyüklüklerine göre ayrıştırılır. Dört reaksiyonun (A, C, G ve T) her birindeki DNA parçaları elektrik alanının etkisiyle jel içinde birbirine paralel ayrı yollardan (bu yollara şerit veya çizgi denir) ilerleyerek ayrıştırılır. Aynı uzunluğa sahip DNA parçları aynı hızda ilerler ve görselleştirildiklerinde (otoradyografi veya mor ötesi ile) birer bant olarak görünürler. Sağdaki resimde jelin üzerine bir röntgen filmi konmuştur, siyah bantlar belli uzunluktaki DNA parçalarına karşılık gelir. Belli bir şeritteki karanlık bant, bir dideoksinükleotitin (ddATP, ddGTP, ddCTP, veya ddTTP) zincire dahi edilmesi sonucu sonlanan bir DNA parçasıdır. Dört şeritteki farklı bantların göreceli konumlarına bakılarak DNA dizisi (alttan yukarı doğru) okunabilir.

Zincir sonlandırma dizilemesinin teknik çeşitlemeleri mevcuttur. Radyoaktif fosfor içeren nükleotitler kullanılarak radyoişaretleme yapılabilir veya 5' ucunda floresan boya ile işaretlenmiş bir primer kullanılabilir. Optik bir sistemde yapılan boya-primer dizilemesi sayesinde okuma daha hızlı ve ekonomik yapılabilir ve sistemin otomatizasyonu mümkün olur. Leroy Hood ve arkadaşları tarafından geliştirilen bu sistemler otomatik ve yüksek hacimli DNA dizilemesini mümkün kıldı.[14][15]

Zincir sonlandırması DNA dizilemesini son derece kolaylaştırmıştır. Örneğin, ticarî olarak elde edilebilen zincir sonladırma kitlerinde dizileme yapmak için gerekli olan reaktantlar, taksim edilmiş ve kullanıma hazır şekilde bulunur. Yöntemin sınırlı kalabileceği durumlar, (1) primerin DNA'ya non-spesifik bağlanıp DNA dizisinin doğru okunmamasına neden olması ve (2) DNA'daki ikincil yapıların dizinin sadakatine etki etmesidir.
Boya sonlandırmalı dizileme

Boya sonlandırmalı dizilemede (İng. Dye-terminator sequencing) zincir sonladırıcı ddNTP'lerin işaretlenir, böylece işaretlenmiş primer yönteminde olduğu gibi dört reaksiyon yerine, dizileme tek bir reaksiyon içinde yürütülebilir. Boya sonladırma dizilemesinde dört dideoksinükleotit bitiricilerin her biri farklı floresan boyalarla işaretlidir, bunların her biri farklı dalga boylarında ışık salar.
Daha az işlem gerektirdiğinden ve daha hızlı çalıştığından dolayı, günümüzde otomatik dizileme makinalarının çoğunda boya sonladırma dizilemesi yapılır. Bu yöntemin bazı sınırlamaları vardır, boya işaretli zincir sonladırıcıların DNA parçasına dahil oluşunda boyadan boyaya farklar mevcuttur, bunun sonucu kapiler elektroforezde elde edilen dizi kromatogramındaki pik yükseklikleri ve şekilleri eşitsizlikler gösterir (soldaki şekle bakınız). Modifiye edilmiş DNA polimeraz enzim sistemleri ve DNA'ya dahil olmadaki çeşitliliği azaltan boyalar ile bu problem çözülmüştür. Boya sonladırma yöntemi ile otomatize edilmiş yüksek hacimli DNA dizi analiz makinaları günümüzde dizileme projelerinin büyük bir çoğunluğunda kullanılmaktadır.
Zorluklar
DNA dizilemesinde sık rastlanan sorunlar arasında ilk 15-40 baz dizisinin kötü kaliteli olması, ve 700-900 bazdan sonra dizileme kromatogramının kalitesinin kötüleşmesidir. Baz bildirim (İng. Base calling) yazılımları kalite kırpması (İng.quality trimming) için tipik olarak bir kalite değeri de verir.[16][17]
DNA parçaları dizilemeden önce klonlanmışsa, elde edilen dizide klonlama vektörüne ait kısımlar da olabilir. Buna karşın, PCR-temelli klonlama ve pirodizilemeye dayalı yeni dizileme teknolojilerinde klonlama vektörü olmaz. Ampliseq ve SeqSharp gibi tek adımlı Sanger dizileme (kombine amplifikasyon ve dizileme) yöntemleri sayesinde hedef genler klonlanmadan ve önceden çoğaltılmadan hızlı dizilenebilmektedir.[18][19]
Mevcut yöntemler tek bir reaksiyonda sadece nispeten kısa (300-1000 nükleotit uzunlukta) DNA parçalarını doğrudan dizileyebilmektedir. Bu limitin üzerindeki DNA parçalarının dizilenmesindeki ana engel, uzunluk farkı bir nükleotit olan büyük DNA parçaları için ayrım gücünün yetersiz olmasıdır.
Otomasyon ve numune hazırlaması

Otomatik DNA dizileme aletleri (DNA dizileyicileri) bir defada 384 DNA numunesini dizileyebilir ve bu işlemi günde 24 kere tekrarlayabilir. DNA dizileyicileri, kapiler elektroforez kullanarak büyüklük ayrıştırması yapar, boya floresansının tespit ve kayıt eder, ve floresan pik çizim kromatogramı olarak veri çıkarır. Termal döndürücü (İng. thermal cycler) ile dizileme reaksiyonlarının ardından temizleme ve bir tampon çözelti içinde yeniden çözeltme, sonra da dizileyiciye yükleme işlemleri ayrı ayrı yapılır. Düşük kaliteli DNA dizilerinin kırpılmasını otomatik olarak yapabilen çeşitli yazılımlar mevcuttur. Bu programlar her pikteki kaliteyi belirleyip, genelde dizinin sonlarında bulunan düşük kaliteli baz piklerini atarlar. Bu programların doğruluk derecesi insan operatörlerinkinden daha düşüktür ama çok büyük veri kümelerinin otomatik işlenmesi için yeterlidir.
Amplifikasyon ve klonal seleksiyon
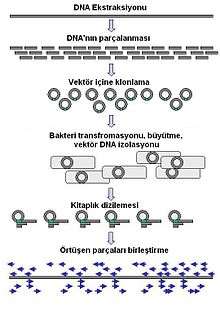
Büyük ölçekli dizilemenin amacı, kromozom gibi çok uzun DNA parçalarını dizilemektir. Yaygın kullanılan yollardan biri büyük DNA parçalarını restriksiyon enzimleri veya mekanik güçler kullanarak küçük parçalara ayırmaktır. Parçalanmış DNA bir DNA vektörü içine klonlanır ve Escherichia coli içinde çoğaltılır. Her bir bakteriyel koloniden saflaştırılan kısa DNA parçaları ayrı ayrı dizilenir ve tek bir uzun, bitişik dizi şeklinde elektronik olarak birleştirilir.
Bu yöntem DNA dizisi hakkında bir ön bilgi gerektirmez ve de novo dizileme olarak adlandırılır. Birleştirilmiş dizideki boşluklar primer yürümesi yapılarak doldurulabilir. Farklı stratejilerin hız ve doğruluk açısından farklı avantajları vardır; saçma yöntemleri (İng. shotgun sequencing) çoğu zaman büyük genomları dizilemekte kullanılır ama elde edilen verinin birleştirmesini yapmak karmaşık ve zordur, özelikle dizi tekrarlarının neden olduğu bölgelerde.
Çoğu dizileme yönteminde bireysel DNA moleküllerinin çoğaltılması için bir in vitro klonlama adımı kullanılır, çünkü moleküler tespit yöntemleri tekil moleküllerin dizilemesine yetecek duyarlıkta değildir. Emülsiyon PCR yönteminde[20] bireysel DNA molekülleri ve primer kaplı boncuklarlar, yağ fazı içinde bulunan sulu damlacıklar içinde izole edilir. Polimeraz zincir tepkimesi (PCR) ile her boncuk DNA molekülünün klonal kopyaları ile kaplanır, sonra bu boncuklar daha sonra dizilemede kullanılmak üzere sabitlenir (immobilize edilir). Emülsiyon PCR Marguilis ve arkadaşları (454 Life Sciences tarafından ticarileştirilmiştir), Shendure ve Porreca ve arkadaşları (Poloni dizilemesi olarak bilinir) ve SOLiD dizilemesi (Agencourt tarafından geliştirilmiştir, artık Applied Biosystems bünyesindedir).[21][22][23]
In vitro klonal çoğaltma için kullanıla bir diğer yöntem köprü PCR (İng. bridge PCR) olarak adlandırılır, bunda DNA parçaları katı bir yüzeye bağlanmış primerler üzerinde çoğaltılır (bu yöntem Illumina Genome Analyzer makinasında kullanılır). Tek molekül yöntemleri (Stephen Quake'in laboratuvarında geliştirilen ve daha sonra Helicos tarafından ticarileştirilen) bir istisnadır: bu yöntemde parlak floresan boyalar ve lazer uyarımı kullanılarak bir yüzey üzerine sabitleştirilmiş bireysel DNA moleküllerindeki baz ekleme olayları tespit edilir, böylece moleküler çoğaltma gereği ortadan kalkar.[24]
Yüksek hacimli dizileme
Düşük masraflı dizilemeye olan büyük talep, yüksek hacimli dizileme teknolojilerinin geliştirilmesine yol açmıştır. Bu teknolojilerde dizileme süreci paralelleştirilerek binlerce veya milyonlarca dizi aynı anda üretilir.[25][26] Yüksek hacimli dizileme teknolojilerinin amacı, standart boya sonlandırma yöntemleri ile mümkün olan DNA dizileme masrafını azaltmaktır.[27]
Lynx Therapeutics'in Kitlesel Paralel İmza Dizilemesi
"Gelecek kuşak" dizileme teknolojilerinin ilki olan Kitlesel Paralel İmza Dizilemesi (İng. Massively Parallel Signature Sequencing veya MPSS), 1990'larda Lynx Therapeutics'te geliştirilmiştir. Bu şirket 1992'de Sydney Brenner ve Sam Eletr tarafından kurulmuştur. MPSS, boncuk temelli bir yöntemdi, bir adaptör ligasyonu ve onu takip eden bir adaptör deşifrasyon adımı içeriyordu, ve dizi dört nükleotitlik birimler halinde okunuyordu. Bu yöntem diziye özgü taraflılığa yol açıyordu ve spesifik dizilerin kaybına yol açıyordu. Teknoloji çok karmaşık olduğu için MPSS sadece Lynx Therapeutics tarafından bir hizmet olarak sunulmaktaydı ve dizileme makinası satılmıyordu. Daha sonradan Solexa ile şirket evliliği olunca bu teknoloji, sentez yoluyla dizilemenin gelişmesine yol açtı, çok daha basit olan bu yeni yaklaşım sayesinde MPSS kulanılmaz oldu. Ancak, MPSS çıktısının esas özellikleri daha sonra gelen "gelecek kuşak" (İng. "next gen") veri tiplerine çok benzemekteydi, yüzlerce bin sayıda kısa diziler üretmesi de dahil olmak üzere. MPSS durumunda, bu kısa diziler tipik olarak cDNA dizilemesinde, gen ifade düzeylerini ölçmek amacıyla kullanılıyordu. Lynx Therapeutics 2004'te Solexa ile birleşti ve bu şirket de daha sonra Illumina tarafından satın alındı.[28]
Poloni dizilemesi
Harvard'da George Church'ün laboratuvarında geliştirilen poloni dizilemesi, 2005'te tüm bir genomu dizilemek için kullanılan ilk gelecek-kuşak dizileme sistemleri arasındaydı. In vitro Eşli etiket ("paired-tag") kitaplığı, emülsiyon PCR'si, otomatik bir mikroskop ve ligasyon-temelli dizileme kimyasını birleştirerek bir E. coli genomunu %99.9999'dan yüksek bir doğruluk üretebilmiş ve bunun maliyetini Sanger dizilemesinin yaklaşık 1/10'una getirmiştir. Bu teknoloji önce Agencourt Biosciences şirketine lisanslanmış, daha sonra ana şirketten ayrılan Agencourt Personal Genomics'e aktarılmış ve en nihayet Applied Biosystems'in SOLiD dizileme platformuna dahil edilmiştir.
454 pirodizilemesi
Pirodizilemenin paralelleştirilmiş bir versiyonu 454 Life Sciences tarafından geliştirilmiştir. Bu yöntemde bir yağ solüsyonu içindeki su damlacıklarında yer alan DNA, PCR ile çoğaltılır (emülsiyon PCR'si). Tek bir DNA kalıbının tek bir primer kaplı boncuğa bağlı olduğu her damlacık, sonra klonal bir koloni oluşturur. Dizileme makinası çok sayıda pikolitre hacimli kuyulara sahiptir, bunların her biri tek bir boncuk ve dizileme enzimleri içerir. Pirodizilemede lüsiferaz enzimi kullanılır, bu enzimin ürettiği ışık ile büyümekte olan DNA'ya eklenen her bir nükleotitin tespit edilir. Veriler birleştirilerek dizi okumaları üretilir.[21] Bu teknoloji orta uzunlukta diziler üretir, baz başına maliyeti Sanger dizilemesi ile SOLiD arasında yer alır.[29]
Solexa dizilemesi
Solexa tersinir boya sonlandırıcılarına dayanan dizileme teknolojisini geliştirmiştir. DNA molekülleri bir lam üzerindeki primerlere bağlanır, sonra çoğaltılarak lokal, klonal koloniler oluştururlar (köprü amplifikasyonu). Dört tip ddNTP eklenir, dahil edilmeyen nükleotitler yıkanıp atılır. Pirodizilemeden farklı olarak, DNA ancak bir defada bir nükleotit kadar uzatılabilir. Bir kamera ile floresan işaretli nükleotitlerin resmi çekilir. Sonra 3' uçtaki bloker ve floresan boya beraberce kimyasal olarak çıkarılır ve bir sonraki döngü başlar.[30]
SOLiD dizilemesi
Applied Biosystems'in SOLiD teknolojisinde ligasyon yoluyla dizileme yöntemi kullanılır. Burada, belli uzunluktaki tüm olası oligonükleotitlerin bir karışımı dizilenen konuma göre işaretlenir. Oligonükleotitler tavlanır ve ligasyona uğrar; DNA ligazın birbiriyle eşleşen dizileri bağlama tercihi nedeniyle, o pozisyon için bilgilendirici bir sinyal elde edilir. Dizilemeden önce DNA emülsiyon PCR ile çoğaltılır. Elde edilen boncukların her birinde aynı DNA'nın kopyaları bulunmaktadır, bu boncuklar bir cam lamın üzerine yerleştirilir.[31] Sonuç, Illumina dizilemesine benzer sayı ve uzunluklarda dizilerdir.[29]
İyon yarı iletken dizilemesi
Ion Torrent Systems Inc. iyon yarı iletken dizilemesine dayalı bir sistem geliştirmiştir. Bu dizileme yöntemi, DNA'nın polimerizasyonu sırasında açığa çıkan hidrojen iyonlarının tespitine dayalıdır. Dizilenecek bir kalıp DNA ipliği içeren bir mikrokuyu, tek bir nükleotit tipi ile doldurulur. Eğer eklenen nükleotit uzayan zincire dahil edilebiliyorsa bu bir hidrojen iyonunun salınmasına yol açar, bu da çok hassas bir iyon sensorunu uyarır. Eğer kalıp diziden aynı nükleotitten peşpeşe birkaç tane varsa (homopolimer tekrar), bir döngüde birden çok nükleotit zincire katılacaktır. Bu durumda daha çok sayıda hidrojen iyonu salınacak ve orantılı olarak daha yüksek bir elektronik sinyal elde edilecektir.[32][33]
DNA nanotop dizilemesi
DNA nanotop dizilemesi Complete Genomics şirketi tarafından geliştirilmiş bir dizileme teknolojisidir. Yöntem, dönen çember ikileşmesi kullanarak genomik DNA'dan elde edilmiş küçük parçaları çoğaltıp bunlardan DNA nanotopları meydana getirmektedir. Ligaz yoluyla dizileme ile nükleotit dizisi belirlenir.[34] Bu yöntem bir defada çok sayıda DNA nanotoplarının dizilenmesini sağlar ve diğer gelecek-kuşak dizileme platformlarına kıyasla daha düşük rektant masrafı vardır.[35] Ancak, her bir DNA nanotopundan sadece kısa diziler elde edilir, bu yüzden bu kısa dizilerin bir referans genomu üzerinde haritalanması zordur.[36] Bu teknoloji çeşitli genom dizileme projelerinde uygulanmıştır.[37]
Gelecek yöntemler
Hibridizasyon yoluyla dizileme DNA mikrodizilimi kullanan non-enzimatik bir yöntemdir. Dizisi belirlenecek olan bir DNA havuzu, floresan işaretlenip üzerinde bilinen diziler bulunan bir mikroçipe hibiridize edilir. Çip üzerinde belli bir noktada kuvvetli hibridizasyon sinyali olması, dizilenmekte olan DNA'da o dizinin bulunduğunu gösterir.[38] Kütle spektrometresi zincir sonlandırma reaksiyonlarında meydana gelen DNA parçaları arasındaki kütle farklarının belirlenmesi için kullanılabilir.[39]
Halen geliştirilmekte olan DNA dizileme yöntemleri arasında DNA polimerazın işaretlenmesi,[40] DNA bir nanopordan geçerken dizisinin okunması,[41][42] ve ağır elementlerle (halojenler gibi) nükleotitleri işaretleyerek uzun DNA parçalarıdaki (>5,000 bp) bireysel nükleotitlerin yerlerini tespit etmeyi amaçlayan mikroskopi temelli teknikler (atomik kuvvet mikroskopu veya transmisyon elektron mikroskopisi ).[43][44]
Ekim 2006'da X Prize Foundation (X Ödül Vakfı) tüm genom dizileme teknolojilerinin geliştirilmesine önayak olmak için Archon X Ödülü'nü ilan etti. Bu ödül "100 insan genomunu 10 gün veya daha kısa bir zamanda, dizilenen her 100.000 bazdan en fazla 1 hatalık bir doğrulukla, genomdaki dizilerin en az %98'ini içeren diziler üreten ve genom başına $10,000 (US) maliyete bunu yapabilen ekibe" $10 milyon ödül vadetmektedir.[45]
Her sene NHGRI genomiks sahasında yeni araştırma ve geliştirmeler için fonlar vermektedir. 2010 ve 2011'de desteklenen çalışmalar mikroakışkan, poloni ve ağır baz dizileme yöntemleri ile ilgiliydi.[46]
DNA dizilemesinde önemli aşamalar
- 1953: DNA çifte sarmal yapısının keşfi.[47]
- 1972: Rekombinant DNA teknolojisinin keşfi. Bunun sayesinde tanımlı DNA parçalarının izolasyonu mümkün hâle gelmiştir. Bundan evvel dizilenebilen numeneler sadece bakteriyofaj veya virüs DNA'larıydı.
- 1977: İlk dizilenen DNA genom bacteriophage φX174.[48]
- 1977: Allan Maxam ve Walter Gilbert "Kimyasal yıkım ile DNA dizilemesi" adlı makalelerini yayımladılar.[5] Bağımsız olarak, Frederick Sanger, "Zincir sonlandırma inhibitörleri ile DNA dizilemesi" adlı makalesini yayımlıyor.[49]
- 1984: Britanya'da Tıbbi Araştırma Derneği araştırmacıları Epstein-Barr virüsünün DNA dizisini (170 kb) yayımladılar .
- 1986: California Institute of Technology'de Leroy E. Hood'un laboratuvarı ve Smith ilk yarı otomatik DNA dizileme makinasını ilan ettiler.
- 1987: Applied Biosystems ilk tamamen otomatik otomatik dizileme makinası olan model ABI 370'ı pazarladı.
- 1990: ABD Millî Sağlık kurumu (NIH), Mycoplasma capricolum, Escherichia coli, Caenorhabditis elegans, ve Saccharomyces cerevisiae için ilk büyük ölçekli denemelere başladı (baz başına US$0.75 masraf ile).
- 1991: ifade edilmiş dizi etiketlerinin (İng. expressed sequence tag) dizilenmesi Craig Venter'in laboratuvarında başladı, insan genomunun kodlayıcı kısmını elde etmek amacıyla.[50]
- 1995: The Institute for Genomic Research (TIGR)'da Craig Venter, Hamilton Smith, ve arkadaşları doğada yaşayan bir organizmanın (Haemophilus influenzae bakterisinin) ilk genom dizisini yayımladılar. Dairesel kromozomda 1.830.137 baz bulunmaktaydı ve Science dergisinde yayımlanması[51] tüm-genom saçma dizilime (whole-genome shotgun sequencing) yönteminin ilk kullanımı olmuştur, bu yöntem ile, daha evvelki dizileme projelerinde kullanılan baştan bir haritalama aşaması bertaraf edilmiştir.
- 1996 Stokholm'daki Kraliyet Teknoloji Enstitüsü'nden Pål Nyrén ve öğrencisi Mostafa Ronaghi pirodizileme yöntemini yayınladılar[52]
- 1998 University of Washington'dan Phil Green ve Brent Ewing, dizileyici veri analizi için
“phred”yayımladılar.[53] - 2000: Lynx Therapeutics, paralelleştirilmiş, adaptör/ligasyon aracılıklı, boncuk tabanlı bir dizileme teknolojisi olan "MPSS"yi yayımladı ve pazarlayıp "gelecek kuşak" dizileme akımını başlattı.[54]
- 2001: insan genomunun taslak sürümü yayımlandı.[55][56]
- 2004: 454 Life Sciences pirodizilemenin paralelleştirilmiş bir versiyonunu pazarladı.[57][58] Makinalarının ilk sürümü, otomatik Sanger dizilemesine kıyasla dizileme masraflarını 6-kat azaltmakta, ve MPSS'ninkinin ardından "gelecek kuşak" dizileme makinalarının ikincisiydi.[29]
Kaynaklar
- ↑ Olsvik O, Wahlberg J, Petterson B, et al. (January 1993). "Use of automated sequencing of polymerase chain reaction-generated amplicons to identify three types of cholera toxin subunit B in Vibrio cholerae O1 strains". J. Clin. Microbiol. 31 (1): 22–5. PMC 262614. PMID 7678018. http://jcm.asm.org/cgi/pmidlookup?view=long&pmid=7678018.
- ↑ Pettersson E, Lundeberg J, Ahmadian A (February 2009). "Generations of sequencing technologies". Genomics 93 (2): 105–11. DOI:10.1016/j.ygeno.2008.10.003. PMID 18992322.
- ↑ Min Jou W, Haegeman G, Ysebaert M, Fiers W (May 1972). "Nucleotide sequence of the gene coding for the bacteriophage MS2 coat protein". Nature 237 (5350): 82–8. DOI:10.1038/237082a0. PMID 4555447.
- ↑ Fiers W, Contreras R, Duerinck F, et al (April 1976). "Complete nucleotide sequence of bacteriophage MS2 RNA: primary and secondary structure of the replicase gene". Nature 260 (5551): 500–7. DOI:10.1038/260500a0. PMID 1264203.
- 1 2 3 Maxam AM, Gilbert W (February 1977). "A new method for sequencing DNA". Proc. Natl. Acad. Sci. U.S.A. 74 (2): 560–4. DOI:10.1073/pnas.74.2.560. PMC 392330. PMID 265521. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=392330.
- ↑ Gilbert, W. DNA sequencing and gene structure. Nobel lecture, 8 December 1980.
- ↑ Gilbert W, Maxam A (December 1973). "The nucleotide sequence of the lac operator". Proc. Natl. Acad. Sci. U.S.A. 70 (12): 3581–4. DOI:10.1073/pnas.70.12.3581. PMC 427284. PMID 4587255. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=427284.
- 1 2 Sanger F, Coulson AR (May 1975). "A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase". J. Mol. Biol. 94 (3): 441–8. DOI:10.1016/0022-2836(75)90213-2. PMID 1100841.
- ↑ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proc. Natl. Acad. Sci. U.S.A. 74 (12): 5463–7. DOI:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=431765.
- ↑ Sanger F. Determination of nucleotide sequences in DNA. Nobel lecture, 8 December 1980.
- ↑ Graziano Pesole; Cecilia Saccone (2003). Handbook of comparative genomics: principles and methodology. New York: Wiley-Liss. s. 133. ISBN 0-471-39128-X. http://books.google.com/books?id=dXk0JvN2Y-IC&pg=PA133.
- ↑ "Cold Spring Harb Protoc -- Sign In Page". 25 Temmuz 2011 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20110725210644/http://cshprotocols.cshlp.org/cgi/content/full/2006/1/pdb.prot3804?ijkey=17dc0499c9e0aa3b22887d5332698df00217774f&keytype2=tf_ipsecsha.
- ↑ "Cold Spring Harb Protoc -- Sign In Page". 25 Temmuz 2011 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20110725211556/http://cshprotocols.cshlp.org/cgi/content/full/2007/12/pdb.prot4812.
- ↑ Smith LM, Sanders JZ, Kaiser RJ, et al (1986). "Fluorescence detection in automated DNA sequence analysis". Nature 321 (6071): 674–9. DOI:10.1038/321674a0. PMID 3713851. "We have developed a method for the partial automation of DNA sequence analysis. Fluorescence detection of the DNA fragments is accomplished by means of a fluorophore covalently attached to the oligonucleotide primer used in enzymatic DNA sequence analysis. A different coloured fluorophore is used for each of the reactions specific for the bases A, C, G and T. The reaction mixtures are combined and co-electrophoresed down a single polyacrylamide gel tube, the separated fluorescent bands of DNA are detected near the bottom of the tube, and the sequence information is acquired directly by computer."
- ↑ Smith LM, Fung S, Hunkapiller MW, Hunkapiller TJ, Hood LE (April 1985). "The synthesis of oligonucleotides containing an aliphatic amino group at the 5' terminus: synthesis of fluorescent DNA primers for use in DNA sequence analysis". Nucleic Acids Res. 13 (7): 2399–412. DOI:10.1093/nar/13.7.2399. PMC 341163. PMID 4000959. http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=4000959.
- ↑ "Phred - Quality Base Calling". 26 Nisan 2015 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20150426090626/http://www.phrap.com:80/phred/. Erişim tarihi: 2011-02-24.
- ↑ "Base-calling for next-generation sequencing platforms — Brief Bioinform". 17 Haziran 2015 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20150617164901/http://bib.oxfordjournals.org:80/content/early/2011/01/18/bib.bbq077.full. Erişim tarihi: 2011-02-24.
- ↑ DOI:10.1373/clinchem.2004.039164
Bu alıntı, sonraki birkaç dakika içinde otomatik olarak tamamlanacaktır. Siz de kuyruğun önüne geçebilir ya da elle açıklayabilirsiniz - ↑ DOI:10.2353/jmoldx.2010.090134
Bu alıntı, sonraki birkaç dakika içinde otomatik olarak tamamlanacaktır. Siz de kuyruğun önüne geçebilir ya da elle açıklayabilirsiniz - ↑ Richard Williams, Sergio G Peisajovich, Oliver J Miller, Shlomo Magdassi, Dan S Tawfik, Andrew D Griffiths (2006). "Amplification of complex gene libraries by emulsion PCR". Nature methods 3 (7): 545–550. DOI:10.1038/nmeth896. PMID 16791213.
- 1 2 Margulies M, Egholm M, Altman WE, et al (September 2005). "Genome sequencing in microfabricated high-density picolitre reactors". Nature 437 (7057): 376–80. DOI:10.1038/nature03959. PMC 1464427. PMID 16056220. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1464427.
- ↑ Shendure, J.; Porreca, GJ; Reppas, NB; Lin, X; McCutcheon, JP; Rosenbaum, AM; Wang, MD; Zhang, K ve diğ. (2005). "Accurate Multiplex Polony Sequencing of an Evolved Bacterial Genome". Science 309 (5741): 1728. DOI:10.1126/science.1117389. PMID 16081699.
- ↑ Applied Biosystems' SOLiD technology
- ↑ Braslavsky I, Hebert B, Kartalov E, Quake SR (April 2003). "Sequence information can be obtained from single DNA molecules". Proc. Natl. Acad. Sci. U.S.A. 100 (7): 3960–4. DOI:10.1073/pnas.0230489100. PMC 153030. PMID 12651960. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=153030.
- ↑ Hall N (May 2007). "Advanced sequencing technologies and their wider impact in microbiology". J. Exp. Biol. 210 (Pt 9): 1518–25. DOI:10.1242/jeb.001370. PMID 17449817.
- ↑ Church GM (January 2006). "Genomes for all". Sci. Am. 294 (1): 46–54. DOI:10.1038/scientificamerican0106-46. PMID 16468433.
- ↑ Schuster, Stephan C. (2008). "Next-generation sequencing transforms today's biology". Nature methods (Nature Methods) 5 (1): 16–18. DOI:10.1038/nmeth1156. PMID 18165802.
- ↑ Brenner, Sidney; Johnson, M; Bridgham, J; Golda, G; Lloyd, DH; Johnson, D; Luo, S; McCurdy, S ve diğ. (2000). "Gene expression analysis by massively parallel signature sequencing (MPSS) on microbead arrays". Nature Biotechnology (Nature Biotechnology) 18 (6): 630–634. DOI:10.1038/76469. PMID 10835600.
- 1 2 3 Schuster SC (January 2008). "Next-generation sequencing transforms today's biology". Nat. Methods 5 (1): 16–8. DOI:10.1038/nmeth1156. PMID 18165802.
- ↑ Mardis ER (2008). "Next-generation DNA sequencing methods". Annu Rev Genomics Hum Genet 9: 387–402. DOI:10.1146/annurev.genom.9.081307.164359. PMID 18576944.
- ↑ Valouev A, Ichikawa J, Tonthat T, et al. (July 2008). "A high-resolution, nucleosome position map of C. elegans reveals a lack of universal sequence-dictated positioning". Genome Res. 18 (7): 1051–63. DOI:10.1101/gr.076463.108. PMC 2493394. PMID 18477713. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2493394.
- ↑ Rusk, N. (2011). "Torrents of sequence." Nat Meth 8(1): 44-44.
- ↑ Ion Torrent Official Webpage.
- ↑ Human Genome Sequencing Using Unchained Base Reads in Self-Assembling DNA Nanoarrays. Drmanac, R. et. al. Science, 2010, 327 (5961): 78-81,
- ↑ Genome Sequencing on Nanoballs Porreca, JG. Nature Biotechnology, 2010, 28:(43-44)
- ↑ Human Genome Sequencing Using Unchained Base Reaads in Self-Assembling DNA Nanoarrays, Supplementary Material. Drmanac, R. et. al. Science, 2010, 327 (5961):78-81,
- ↑ Complete Genomics Press release, 2010
- ↑ Hanna GJ, Johnson VA, Kuritzkes DR, et al (1 July 2000). "Comparison of sequencing by hybridization and cycle sequencing for genotyping of human immunodeficiency virus type 1 reverse transcriptase". J. Clin. Microbiol. 38 (7): 2715–21. PMC 87006. PMID 10878069. http://jcm.asm.org/cgi/pmidlookup?view=long&pmid=10878069.
- ↑ J.R. Edwards, H.Ruparel, and J. Ju (2005). "Mass-spectrometry DNA sequencing". Mutation Research 573 (1-2): 3–12. DOI:10.1016/j.mrfmmm.2004.07.021. PMID 15829234.
- ↑ "VisiGen Biotechnologies Inc. - Technology Overview". Visigenbio.com. 7 Eylül 2008 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20080907134124/http://visigenbio.com/technology_overview.html. Erişim tarihi: 2009-11-15.
- ↑ "The Harvard Nanopore Group". Mcb.harvard.edu. 4 Haziran 2012 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20120604203545/http://mcb.harvard.edu:80/branton/index.htm. Erişim tarihi: 2009-11-15.
- ↑ "Nanopore Sequencing Could Slash DNA Analysis Costs". 21 Kasım 2011 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20111121183402/http://www.physorg.com/news157378086.html.
- ↑ US patent 20060029957, ZS Genetics, "Systems and methods of analyzing nucleic acid polymers and related components", çıkış tarihi: 2005-07-14
- ↑ Xu M, Fujita D, Hanagata N (December 2009). "Perspectives and challenges of emerging single-molecule DNA sequencing technologies". Small 5 (23): 2638–49. DOI:10.1002/smll.200900976. PMID 19904762.
- ↑ "PRIZE Overview: Archon X PRIZE for Genomics"
- ↑ The Future of DNA Sequencing
- ↑ Watson JD, Crick FH (1953). "The structure of DNA". Cold Spring Harb. Symp. Quant. Biol. 18: 123–31. PMID 13168976.
- ↑ Sanger F, Air GM, Barrell BG, et al. (February 1977). "Nucleotide sequence of bacteriophage phi X174 DNA". Nature 265 (5596): 687–95. DOI:10.1038/265687a0. PMID 870828.
- ↑ Sanger F, Nicklen S, Coulson AR (December 1977). "DNA sequencing with chain-terminating inhibitors". Proc. Natl. Acad. Sci. U.S.A. 74 (12): 5463–7. DOI:10.1073/pnas.74.12.5463. PMC 431765. PMID 271968. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=431765.
- ↑ Adams MD, Kelley JM, Gocayne JD, et al. (June 1991). "Complementary DNA sequencing: expressed sequence tags and human genome project". Science 252 (5013): 1651–6. DOI:10.1126/science.2047873. PMID 2047873.
- ↑ Fleischmann RD, Adams MD, White O, et al. (July 1995). "Whole-genome random sequencing and assembly of Haemophilus influenzae Rd". Science 269 (5223): 496–512. DOI:10.1126/science.7542800. PMID 7542800. http://www.sciencemag.org/cgi/pmidlookup?view=long&pmid=7542800.
- ↑ M. Ronaghi, S. Karamohamed, B. Pettersson, M. Uhlen, and P. Nyren (1996). "Real-time DNA sequencing using detection of pyrophosphate release". Analytical Biochemistry 242 (1): 84–9. DOI:10.1006/abio.1996.0432. PMID 8923969.
- ↑ Ewing B, Green P (March 1998). "Base-calling of automated sequencer traces using phred. II. Error probabilities". Genome Res. 8 (3): 186–94. DOI:10.1101/gr.8.3.186 (inactive 2010-01-07) . PMID 9521922. http://www.genome.org/cgi/pmidlookup?view=long&pmid=9521922.
- ↑ Brenner S, et al. (2000). "Gene expression analysis by massively parallel signature sequencing (MPSS) on microbead arrays". Nature Biotechnology (Nature Biotechnology) 18 (6): 630–634. DOI:10.1038/76469. PMID 10835600.
- ↑ Lander ES, Linton LM, Birren B, et al. (February 2001). "Initial sequencing and analysis of the human genome". Nature 409 (6822): 860–921. DOI:10.1038/35057062. PMID 11237011.
- ↑ Venter JC, Adams MD, Myers EW, et al. (February 2001). "The sequence of the human genome". Science 291 (5507): 1304–51. DOI:10.1126/science.1058040. PMID 11181995.
- ↑ Stein RA (1 September 2008). "Next-Generation Sequencing Update". Genetic Engineering & Biotechnology News 28 (15). http://www.genengnews.com/gen-articles/next-generation-sequencing-update/2584/.
- ↑ Margulies M, Egholm M, Altman WE, et al. (September 2005). "Genome sequencing in microfabricated high-density picolitre reactors". Nature 437 (7057): 376–80. DOI:10.1038/nature03959. PMC 1464427. PMID 16056220. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1464427.